- Редагування геному CRISPR/Cas9
- Короткий огляд
- Вступ
- Історія відкриття CRISPR/Cas9
- Що таке CRISPR/Cas9?
- Механізм роботи системи CRISPR/Cas9
- Хто відкрив систему CRISPR/Cas9?
- Чому CRISPR/Cas9 революційна технологія?
- Інші інструменти для редагування геному
- Системи редагування геному TALEN та ZFN
- Доставка компонентів CRISPR/Cas9 в клітини
- Застосування CRISPR/Cas9 у різних організмах
- Регулювання CRISPR-модифікованих організмів
- Чому відкриття технології CRISPR/Cas9 викликало такий ажіотаж?
- CRISPR як інструмент фундаментальних досліджень
- CRISPR-інтерференція та редагування РНК
- Визнання та нагороди
- Редагування геному людини та етичні проблеми
- Перші експерименти на ссавцях
- Перспективи та проблеми
- Етичні дилеми
- Міжнародні рекомендації щодо редагування генів людини
- Евгеніка та генні мутанти
- Безпека та потенціальні ризики
- Бiологічна зброя та національна безпека
- Біохакінг та домашні експерименти
- Список использованной литературы [REFERENCES]
Редагування геному CRISPR/Cas9
Короткий огляд
В огляді йдеться про історію відкриття, бурхливий розвиток і подальші
перспективи застосування нового потужного інструменту для редагування геному — CRISPR/Cas9. Взявши за основу один з елементів захисної системи бактерій, вчені-біологи створили досить простий, дешевий і швидкий
метод внесення змін у ДНК рослин, тварин і людини. Ніколи раніше людство не мало настільки точного знаряддя для маніпуляції генами, і це відкриває широкі можливості для профілактики та лікування багатьох захворювань. Водночас у суспільстві точаться гострі дискусії: благо чи зло
несе людству CRISPR/Cas9? Як і будь-яка нова технологія, генне редагування викликає побоювання і піднімає низку серйозних етичних проблем,
особливо щодо можливості його використання на клітинах зародкової лінії
і геномі ембріонів людини. Проте вже зараз очевидно, що CRISPR/Cas9 —
це не чергова модна «іграшка» для вчених, а революційна технологія, яка
змінить наше майбутнє.
Стаття (рос): РЕДАКТИРОВАНИЕ ГЕНОМА, ИЛИ CRISPR/CAS9 — ПАНАЦЕЯ ОТ МНОГИХ НЕИЗВОЛЬНЫХ БОЛЕЗНЕЙ ИЛИ ПЕРВЫЙ ШАГ К ГЕННОМУ АПОКАЛИПСИСУ?
Стаття (укр): РЕДАГУВАННЯ ГЕНОМУ, АБО CRISPR/CAS9 — ПАНАЦЕЯ ВІД БАГАТЬОХ НЕВИЛІКОВНИХ ХВОРОБ ЧИ ПЕРШИЙ КРОК ДО ГЕННОГО АПОКАЛІПСИСУ?
Ключові слова: CRISPR/Cas9, редагування геномної ДНК, генна терапія, генетично модифіковані організми.
Автори статьи:
КОМИСАРЕНКО Сергей Васильевич — академик НАН Украины, директор Института биохимии А.В. Палладина НАН Украины;
РОМАНЮК Светлана Ивановна — кандидат биологических наук, старший научный сотрудник Института биохимии А.В. Палладина НАН Украины.
Странца авторов →
Вступ
Історія науки знає численні приклади, коли випадкове відкриття в непопулярній науці призводило до наукового прориву, що робило можливим здійснення найзаповітніших мрій людства. Так були відкриті антибіотики, рентгенівське випромінювання та ін. Ось і зараз ми є свідками розгортання подібної наукової революції в генетиці. Здається, у генетиків з’явився інструмент, здатний у короткі терміни зробити науковою фантастику реальністю. Наукова спільнота активно обговорює перспективи лікування раку та невиліковних спадкових захворювань шляхом видалення з організму дефектних генів. У геометричній прогресії зростає кількість наукових статей на цю тему, у пресі постійно з’являються повідомлення, інтерв’ю, ведуться дискусії.
Винуватцями такого «переполоху» є автори неочікуваного відкриття, які у 2012 році розробили метод CRISPR/Cas9 для точного редагування геному живих організмів.
Відкриття CRISPR/Cas9 вже відзначене чисельними науковими нагородами і, безперечно, заслуговує найпрестижнішу наукову награду — Нобелівську премію. Основна частина цієї статті була написана кілька років тому як передвісник цієї événемента. Однак Нобелівський комітет з різних причин поки не поспішає приймати позитивне рішення, в тому числі, вірогідно, через судовий спір щодо патентування цієї методики, який продовжується останні роки. Попри це, практичне застосування технології CRISPR/Cas9 з кожним роком стає все більш широким і має численні досягнення в галузі біотехнології та медицини, значення яких для людства важко переоцінити. Тому, на нашу думку, важливо та цікаво розглянути основні напрямки та перспективи розвитку цієї технології, а також позначити існуючі проблеми.
Історія відкриття CRISPR/Cas9
З чого ж розпочалася історія відкриття CRISPR/Cas9? Як не дивно, з досліджень протривірусного імунітету бактерій. У 50-х роках XX століття учені звернули увагу на те, що деякі штами бактерій легко заражаються вірусами, а інші штами того ж виду — стійкі до заразу. Двадцять років досліджень дали свої результати: з’ясувалося, що стійкість певних бактеріальних штамів до вірусів пояснюється наявністю у бактерій спеціальних ферментів — рестриктаз (від лат. restrictio — обмеження), які розрізають вірусну ДНК.
Цікавою особливістю цих ферментів є їх чітка специфічність до певної невеликої послідовності ДНК. Власну ДНК бактерія захищає від рестриктаз за допомогою метилування нуклеотидних залишків аденину та цитозину. Першу рестриктазу (EcoK) у 1968 році виділили Метью Мезельсон (Matthew Meselson) та Роберт Юань (Robert Yuan) [1]. Здатність цих ферментів «різати» ДНК в чітко визначеній точці була використана вченими для отримання потрібних фрагментів геномної ДНК. У 1978 році за відкриття рестриктаз та їх застосування в молекулярній генетиці Вернеру Арберу (Werner Arber), Гамільтону Сміту (Hamilton O. Smith) та Даніелю Натансу (Daniel Nathans) була присуджена Нобелівська премія. А у 1967 році Бернард Вейс (Bernhard Weiss) та Чарльз Річардсон (Charles Richardson) вперше виявили спеціальні «зшиваючі» ферменти — ДНК-лігази [2]. Використання лігаз та рестриктаз дозволило створювати штучні конструкції з ДНК живих організмів. Так з’явилася нова галузь науки — генна інженерія, яка дала можливість створювати генно-модифіковані організми (ГМО), рекомбінантні протеїни, індуковані стовбурові клітини та ін.
Що таке CRISPR/Cas9?
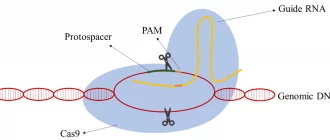
І що це має відношення до протривірусного імунітету бактерій? З’ясувалося, що бактерії захищаються від вірусів не тільки за допомогою рестриктаз, оскільки вірус може не мати у своїй ДНК послідовності, необхідної для розщеплення цими ферментами. Існує й інший, більш ефективний механізм, який забезпечує бактеріям специфічний захист після зустрічі з певним вірусом. Цей механізм реалізує система з досить громіздкою назвою CRISPR/Cas9, або «крісперу».
Коли ДНК вірусу проникає в бактерію, відбувається копіювання фрагмента цієї ДНК та перенесення його у спеціальне «сховище» інформації про віруси у власному геномі — CRISPR (Clustered Regularly Interspaced Short Palindromic Repeats — короткі палідромні повтори, регулярно розташовані групами). Тут зразки ДНК різних вірусів (спейсери) накопичуються між однаковими короткими повторами бактеріальної ДНК та використовуються для виготовлення CRISPR РНК — crРНК (їх також називають РНК-зондами або РНК-гідами), які специфічно розпізнають гени певних вірусів та зв’язуються з ними при повторній заразі. Завдяки crРНК вірусну ДНК знаходять спеціальні ферменти — протеїни Cas (CRISPR-асоційовані протеїни), гени яких розташовані поруч з масивом CRISPR. Ці ферменти, як і рестриктази, є ендонуклеазами, тобто вони можуть розрізати ДНК вірусу, знищуючи її. Розглянемо детальніше, як саме працює система CRISPR/Cas9.
Механізм роботи системи CRISPR/Cas9
Розглянемо більш детально, як саме працює система CRISPR/Cas9. Лідерна послідовність CRISPR грає роль промотора, з якого розпочинається транскрипція всього масиву. Можливо, вона також розпізнається протеїнами, які вставляють в CRISPR нові спейсери. Після транскрипції масиву CRISPR утворюється довга молекула РНК, до якої приєднується невеличка РНК, комплементарна повторам (tracrРНК); до tracrРНК приєднується протеїн Cas9, який активується при взаємодії, а до Cas9, у свою чергу, приєднується фермент РНКаза III, яка розрізає довгу РНК на фрагменти — crРНК та відокремлюється. У Streptococcus pyogenes, наприклад, crРНК, які утворилися, складаються з 42 нуклеотидів: 22 кінцевих нуклеотидів — це половина палідромного повтора, а решта 20 — унікальний спейсерний фрагмент. По суті в результаті розрізання довгої РНК утворюється комплекс crРНК, tracrРНК та активованого протеїну Cas9. Спейсерна частина crРНК розпізнає комплементарну ділянку вірусної ДНК, а протеїн Cas9 закріплюється на двох нитях ДНК та «розрізає» їх, викликаючи подальшу деградацію чужої ДНК [3].
Для успішного розпізнавання ДНК-«мішеней» та її пошкодження необхідно, щоб протеїн Cas9 розпізнавав певну коротку (3-9 нуклеотидів) послідовність PAM (Protospacer Adjacent Motif — мотив, який прилягає до протоспейсера), яка знаходиться поруч з розпізнаною crРНК частиною ДНК. У різних видах бактерій ця послідовність різна; наприклад, для Streptococcus pyogenes послідовність PAM — 5′-NRG3′ (де N — будь-який нуклеотид, а R — A або G).
Для чого потрібне розпізнавання PAM? Є припущення, що відсутність послідовностей PAM у складі власних генів бактерії захищає їх від розрізання системою CRISPR/Cas9, так як CRISPR містить 20% спейсерів, спрямованих на власну ДНК [4]. Чому ці спейсери зберігаються, невідомо. Можливо, у бактерій є якийсь механізм регуляції транскрипції власних генів, в який залучені CRISPR та спеціальні білки.
Хто відкрив систему CRISPR/Cas9?
Суперечки з цього питання тривають і до сьогодні. Адже у сучасній науці вже неможливо щось відкрити односторонньо. Історія відкриття, яка викликала революцію в генній інженерії, тривала впродовж 25 років, і багато учених внесли в неї свій внесок. У 1987 році японські учені під керівництвом Йосідзумі Ішино (Yoshizumi Ishino) разом з геном iap, який вони досліджували, випадково клонували досить великий фрагмент геному кишкової палички Escherichia coli, який не кодував жодного білка [5].
Це було дуже дивно, так як бактерії, як правило, не мають зайвих послідовностей. Ділянка складалася з повторюваних послідовностей ДНК та варіабельних фрагментів ДНК між ними — спейсерів [6]. У 1993 році учені з Нідерландів під керівництвом Яна ван Ембдена (Jan D.A. van Embden) досліджували різноманітність перривистих прямих повторів серед різних штамів збудника туберкульозу (Mycobacterium tuberculosis) та розробили новий метод диференціації штамів, який використовується і по сей день [7].
Пізніше, іспанець Франсіско Хуан Мартінес Мохіка (Francisco Juan Martínez Mojica) виявив подібні ділянки в інших видах бактерій та архей, і у 2000 році запропонував об’єднати їх в сім’ю під назвою SRSRs (Short Regularly Spaced Repeats — короткі регулярно розташовані повтори) [8], яку через два роки перейменували на CRISPR. Тоді ж, у 2002 році, група учених з Нідерландів на чолі з Руудом Янсеном (Ruud Jansen) відкрили гени протеїнів Cas, розташовані поруч з ділянкою CRISPR [9]. А у 2005 році одразу три дослідницькі групи незалежно одна від одної з’ясували, що між повторами CRISPR часто знаходяться послідовності, ідентичні послідовностям ДНК вірусів-бактеріофагів та плазмід [10-12]. Ці результати та припущення про виконання CRISPR функції протривірусного захисту не викликали тоді особливого інтересу. Все змінила наукова стаття, яка вийшла у журналі Science у 2007 році [13].
Її авторами була група учених під керівництвом Філіппа Хорвата (Philippe Horvath) з компанії Danisco (США) — виробника відомих йогуртів Danone (у 2011 році Danisco придбала компанія DuPont за $6,3 млрд). Справа в тому, що при виробництві кисломолочних продуктів існує небезпека заразу молочнокислих бактерій вірусами-бактеріофагами, що може привести до величезних збитків. Тому в компанії Danisco вирішили відібрати для виробництва клони молочнокислих бактерій, стійких до вірусу. А оскільки компанія використовувала CRISPR для класифікації своїх комерційних штамів, учені відразу помітили, що в ділянках CRISPR відібраних клонів з’являлися нові спейсери, які були ідентичні ділянкам вірусного геному.
Дослідники штучно вставили спейсер з послідовністю ДНК вірусу в ділянку CRISPR бактерії, що одразу ж зробило її стійкою до вірусу.
У 2007-2008 рр. були відкриті важливі деталі механізму роботи CRISPR. Група під керівництвом Джона ван дер Уста (John van der Oost), наприклад, показала, що захист CRISPR реалізується через малі РНК [14]. Лучіано Марраффіні (Luciano A. Marraffini) та Ерік Зонтгеймер (Erik J. Sontheimer) з Північно-Західного університету в Еванстоні (штат Іллінойс, США) довели, що система CRISPR спрямована на розрізання ДНК [15]. Вони першими висловили думку про можливість використання CRISPR для редагування геному в гетерологічних системах та навіть подали заявку на патент, але через недостатність експериментального підтвердження в кінцевому підсумку відмовилися від нього [16].
Потім стало відомо, що для виконання захисної функції CRISPR необхідна взаємодія з багатьма протеїнами Cas, одні з яких зв’язуються з малими РНК, інші розпізнають ДНК вірусу, а треті її розрізають. Однак французька дослідниця Еммануель Шарпентьє (Emmanuelle Charpentier), яка працює в основному в Німеччині та Швеції, виявила різновид системи CRISPR, яка для повноцінного захисту вимагала тільки одного дуже великого протеїну Cas — його назвали Cas9. У 2012 р. Мартін Джінек (Martin Jinek) з групи Дженніфер Дудни (Jennifer Doudna) з Каліфорнійського університету в Берклі (University of California, Berkeley) об’єднав tracrРНК та crРНК в одну єдину молекулу sgРНК (single-guide RNA — єдина РНК-гід) та створив вектор для клонування цієї РНК. З’ясувалося, що така синтетична sgРНК коректно працює в клітині в комплексі з протеїном Cas9.
Тоді наукові групи Е. Шарпентьє та Дж. Дудни опублікували статтю в Science [17], в якій запропонували спосіб використання системи CRISPR/Cas9 для розрізання вибраних дослідником ДНК-«мішеней» в клітині та продемонстрували можливість такого підходу в експериментах in vitro. Майже одночасно з ними подібну статтю опублікувала група литовського біохіміка Вірджініуса Шиксніса (Virginijus Siksnys) з Інституту біотехнології Вільнюського університету [18]. Цікаво, що робота Шиксніса була виконана раніше, але її публікація затрималася на півроку через необґрунтоване відхилення у журналі Cell.
На початку 2013 року групи Джорджа Черча (George Church) [19] та його колишнього аспіранта Фена Чжана (Feng Zhang) [20] з Інституту Broad (Broad Institute of MIT and Harvard) в Кембриджі показали, що штучна crРНК та протеїн Cas9 повноцінно функціонують в клітинах вищих організмів. Практично одночасно ефективність такого підходу підтвердили учені з Південної Кореї в експериментах з культурою клітин людини [21]. Одразу після цього всі забули про проблеми бактерій в їх непростій боротьбі з вірусами. Здавалося, що наукові та громадські видання «вибухнули» публікаціями виключно на одну тему: про CRISPR/Cas9 як фантастичний інструмент для редагування геномів вищих організмів.
Чому CRISPR/Cas9 революційна технологія?
Річ у тому, що ця технологія виявилася набагато кращою за попередні методи генної інженерії. Раніше при створенні ГМО учені не мали можливості контролювати, куди саме у геномі та в якій кількості вбудовується вірусний вектор з геном-вставкою. Щоб отримати необхідний результат, потрібно було провести дуже важку та довготривалу роботу, відкидаючи значну кількість невдалих варіантів. Технологія CRISPR/Cas9 запропонувала простий спосіб вбудовування гена-вставки в певну послідовність ДНК. Крім того, ця технологія дозволяє працювати в клітинах всіх організмів — від бактерій до ссавців. Систему CRISPR/Cas9 можна використовувати не тільки в пробірці, як рестриктази, але й безпосередньо в живій клітині. Можна одночасно редагувати необмежену кількість генів у геномі, а також вводити компоненти системи не тільки в окремі клітини, але й у цілі тканини або весь організм. Безперечні переваги CRISPR/Cas9 значно скорочують тривалість генної інженерії та дозволяють вже зараз розглядати реальну можливість застосування цієї системи для модифікації ембріонів людини, лікування раку або спадкових захворювань.
Інші інструменти для редагування геному
Звісно, існують й інші інструменти для редагування геному, а саме: мегануклеази, TALEN та нуклеази з «цинковими пальцями» (ZFN — Zinc-Finger Nucleases). Мегануклеази — це природні ДНК-нуклеази, які є у фагів, бактерій, грибів, водоростей та деяких рослин. Вони кодуються мобільними генетичними елементами та розпізнають великі ділянки ДНК (12-40 пар основ), завдяки чому їх вважають найбільш специфічними природними рестриктазами.
З моменту відкриття в 1990-х роках найвідомішими стали мегануклеази I-SceI, I-CreI та I-DmoI сім’ї LAGLIDADG (назва походить від характерної для цих протеїнів послідовності). Ці невеликі протеїни зустрічаються в мітохондріях та хлоропластах еукаріотичних одноклітинних організмів та відомі своїми щільно упакованими тривимірними структурами. Однак мегануклеази не отримали широкого застосування в генній інженерії, так як досить складно підібрати серед них фермент, який розрізав би ДНК в потрібному місці. Зараз учені намагаються створити більш досконалі генно-інженерні мегануклеази та їх гібриди з іншими протеїнами [22].
Системи редагування геному TALEN та ZFN
Системи редагування геному TALEN та ZFN — це генно-інженерні протеїни, які містять домен ДНК-нуклеази, що розрізає ДНК, та домени інших протеїнів, які після модифікації набувають здатність зв’язуватися з певною послідовністю ДНК. До складу протеїнів TALEN входить домен протеїну TALE (Transcription Activator-Like Effector — ефектор, подібний до активатора транскрипції) фітопатогенної бактерії Xanthomonas, а протеїни ZFN містять домен еукаріотичного фактора транскрипції, у структурі якого є від 3 до 6 структур так званих «цинкових пальців».
Однак використання систем TALEN та ZFN для редагування геному вимагає створення окремого штучного протеїну для кожної нової ДНК-«мішені». На відміну від них, система CRISPR використовує універсальний протеїн Cas9, та необхідно змінювати тільки sgRNA. Це набагато простіше та дешевше, так як будь-яку РНК можна легко синтезувати. Ще однією перевагою системи CRISPR/Cas9 перед іншими інструментами редагування геному є можливість її застосування до РНК, що забезпечує більш гнучкі можливості впливу на біохімічні процеси в клітині. Для редагування односланцюгової РНК необхідно додати crRNA, протеїн Cas9 та ДНК-олігонуклеотид, що містить послідовність PAM — PAMmer [23]. Так, у 2016 році було запропоновано за допомогою системи CRISPR/Cas9 стежити за часткою певних РНК в живій клітині без застосування генетичних міток — тегів [24].
Доставка компонентів CRISPR/Cas9 в клітини
Біотехнологічні компанії давно продають вектори для зручної доставки в клітину компонентів системи CRISPR/Cas9 з метою редагування геному. Компанія відкритого обміну Addgene (Кембридж, США) восени 2018 р. зробила великий крок, безкоштовно передавши більше 1 млн плазмід учені по всьому світу, а недавно у них з’явилася нова послуга — розповсюдження вірусних векторів, перш за все лентивірусів та адено-асоційованого вірусу (AAV) [25]. Вектори для CRISPR/Cas9 являють собою кільцеву молекулу ДНК, яка кодує sgRNA та матричну РНК протеїну Cas9. Таким чином, дослідник може розрізати будь-яку послідовність ДНК в клітині, включивши в sgRNA відповідний комплементарний фрагмент. Звісно, необхідно вибрати таку послідовність, яка не повторюється в інших ділянках геному. Потім вектор розмножують в клітинах спеціального лабораторного штаму кишкової палички (де він не зчитується через невідповідність командних сигналів), виділяють з бактерій та трансформують ними клітини, геном яких хочуть змінити.
Застосування CRISPR/Cas9 у різних організмах
Учені всього світу кинулися використовувати технологію CRISPR/Cas9 для редагування геномів вірусів, бактерій, тварин та рослин.
Застосування цієї технології до бактерій дозволило з змішаної культури виділяти окремі види та штами. Успішно були модифіковані дріжджі, плодові мухи, рибки данціо, шовкопряди, жаби. Ін’єкції матричної РНК Cas9 та sgRNA в зародышеві клітини дозволили швидко отримувати генетично модифікованих мишей, щурів, свиней, хорів, коропів та навіть слонів. Отже, відкрилися практично безмежні перспективи створення ГМО для боротьби з хворобами, поліпшення порід сільськогосподарських тварин та сортів рослин, створення моделей для вивчення генетичних захворювань людини та тварин, тестування нових методів лікування та навіть створення домашніх тварин.
Регулювання CRISPR-модифікованих організмів
У багатьох країнах, в тому числі в США, організми, модифіковані за допомогою CRISPR, не віднесені до ГМО, так як вони не містять чужорідної ДНК, і тому можна відносно легко отримати дозвіл на вирощування модифікованих CRISPR-рослин та виробництво з них продуктів харчування. Першими в квітні 2016 року такий дозвіл від Міністерства сільського господарства США отримали шампіньйони садові (Agaricus bisporus), яким за допомогою CRISPR позбавили здатність темніти при механічних пошкодженнях та втрачати товарний вигляд. Модифіковані гриби отримав американський учений Йінон Янг (Yinong Yang) з Університету штату Пенсильванія. З тих пір дозвіл на вирощування було дано ще для 5 організмів, модифікованих CRISPR, в тому числі рицевого посіву (Camelina sativa) — олійної культури, що містить більше омега-3 жирних кислот, а також сорту сої, стійкого до посухи. Невдовзі планується виведення цих продуктів на ринок.
Чому відкриття технології CRISPR/Cas9 викликало такий ажіотаж?
Технологія редагування генів здатна підвищити ефективність сільського господарства та допомогти прогодувати зростаюче населення планети, зменшивши при цьому негативний вплив людини на навколишнє середовище. Дослідники планують вирощувати курей, які не викликають алергії у людини, відновити чисельність медоносних бджіл, які постраждали по всьому світу від хвороб та паразитів. Також пропонують застосовувати CRISPR для контролю статі сільськогосподарських тварин: велика рогата худоба повинна народжуватися тільки бичками, які дають більше м’яса; включення гена зеленого флуоресцентного білка (GFP) у статеві хромосоми курей дозволить забраковувати за світінням в ультрафіолеті яйця, з яких вилупляться петушки, та використовувати їх у виробництві вакцин. Однак, так як при редагуванні геному CRISPR може змінювати ненавмисні гени, висловлюється припущення, що це може вплинути на здоров’я тварин, на склад м’яса та молока. Тому поки що споживачі вимагають обережності в застосуванні нових технологій до тварин, які використовуються у виробництві продуктів харчування [27].
Можливість модифікації геномів екзотичних та малодосліджених тварин викликала «хвилю» масового створення нових модельних організмів. Розробка моделей тварин вимагає величезних затрат часу та грошей. У 2016 році Національний науковий фонд США запустив програму вартістю 24 млн доларів для створення нових модельних організмів. Програма підтримує дослідження геномів видів, вивчення життєвих циклів організмів та розробку протоколів для утримання цих видів у лабораторії. У рамках програми в березні 2019 року за допомогою CRISPR була створена перша генетично модифікована рептилія — коричневий анолис (Anolis sagrei) [28].
CRISPR як інструмент фундаментальних досліджень
Технологія CRISPR стала цінним інструментом фундаментальних досліджень, який дозволяє «вимикати» певні гени та встановлювати їх біологічну функцію в організмі. Розкриття генетичних та молекулярних механізмів, які обумовлюють складні ознаки та поведінку, є особливо важливим для малодосліджених видів тварин. Так, за допомогою CRISPR були ідентифіковані гени, які контролюють узір та колір крил метелика [29], створені модифіковані райдерські мурахи (Ooceraea biroi), які не мають запаху феромонів та не можуть підтримувати складну ієрархію колонії. Технологія CRISPR допомогла вивчити мозкову діяльність головоногих молюсків (гавайського кальмара бобтейла Euprymna scolopes та карликового каракатиці Sepia bandensis) під час зміни узорів на шкірі залежно від зовнішнього середовища. Раніше подібні дослідження не проводилися через відсутність у молюсків кісток та неможливості закріплення електродів на голові тварини [28].
CRISPR-інтерференція та редагування РНК
Інактивувати ген без його пошкодження можна за допомогою CRISPR-інтерференції (CRISPRi). При цьому мутантний протеїн dCas9, у якого не функціонують обидва нуклеазні центри, зв’язується з ДНК-«мішенню» та заважає просуванню РНК-полімерази, що призводить до зупинення транскрипції [30]. Якщо до протеїну dCas9 «прив’язати» домен фактора транскрипції, який збільшує або пригнічує активність генів, то можна безпосередньо впливати на функціонування генів та роботу всього організму. Регулювати активність експресії генів можна також, впливаючи на епігеномні процеси (наприклад, на метилування ДНК або ацетилування гістонів).
Для цього можна використовувати штучні білки, що складаються з dCas9 та каталітичного домену відповідного ферменту (ДНК-метилтрансферази або ацетилтрансферази гістонів) [31]. «Мішені» системи CRISPR/Cas9 можуть бути також довгі некодуючі РНК (lncRNA) або енхансерні РНК (eRNA), які регулюють експресію генів та епігенетичні процеси [32]. Це дуже важливо для дослідження функцій регуляторних РНК та встановлення їх ролі у патогенезі захворювань, так як більш ніж 90% захворювань, пов’язаних з однонуклеотидною заміною, містять цю заміну в некодуючих ділянках ДНК [33]. За допомогою CRISPR/Cas9 можна доставляти до мутантних генів білки, здатні замінити дефектний ген на його здорову копію з іншої хромосоми, що може стати основою нових методів лікування деяких захворювань, наприклад, діабету. А приєднавши до білка dCas9 флуоресцентний білок GFP, можна помітити певну ділянку в хромосомі живої клітини та спостерігати за нею під мікроскопом впродовж клітинного циклу або візуально визначати довжину теломер [34]. У 2019 році в Каліфорнійському університеті в Берклі був створений біосенсор CRISPR-Chip на основі ультрачутливого графену та системи CRISPR/Cas9, який дозволяє впродовж 15 хвилин виявляти певні послідовності ДНК без її ампліфікації з чутливістю 1,7 фМ (1,7 × 10^-15 Моль) [35]. Німецькі учені з Університету Фрайбурга розробили електрохімічний мікрофлюїдний біосенсор, який виявляє до восьми мікроРНК в одному зразку за допомогою CRISPR/Cas13a.
Цей біосенсор має універсальний чіп та легко налаштовується на виявлення будь-якої мікроРНК. Він може бути використаний для ранньої діагностики захворювань людини, так як кластери з 5-6 мікроРНК, як правило, є хорошим біомаркером, зокрема, при хворобі Альцгеймера або різних типах раку. Подібні біосенсори, що використовують різні типи ферменту Cas, можуть спрямовуватися на виявлення специфічних послідовностей у одно- або двохланцюговій ДНК та РНК збудників інфекційних захворювань. Така система, наприклад, здатна визначати кількість РНК коронавірусу, його тип (SARS або Covid19) та навіть диференціювати окремі невідповідності нуклеїнової кислоти [36]. Таким чином, як бачимо, можливості системи CRISPR/Cas9 не обмежуються тільки розщепленням певних послідовностей ДНК. Ця система є універсальним механізмом доставки будь-яких молекул у будь-яку «точку» геному, що відкриває фантастичні можливості для медицини та фундаментальної науки.
Визнання та нагороди
Ученим, які внесли найбільший внесок у відкриття технології CRISPR/Cas9, щорічно пророкують Нобелівську премію, а в очікуванні її нагороджують численними іншими нагородами. Більшість нагород зібрала Дж. Дудна (29 за останні 6 років), серед яких: премія Мілдред Кон по біологічній хімії від Американського товариства біохіміків та молекулярних біологів (2013), премія Лурія в галузі біомедичних наук від Фонду Національного інституту здоров’я, премія «Прорив» в галузі наук про життя, премія доктора Поля Янссена по біомедичним дослідженням, премія Якова Гесхеля Габбая (2014), нагорода принцеси Астурійської, премія Грубера в галузі генетики (2015), премія фонду Ворена Альперта, премія Павла Еріліха та Людвіга Дармштедтера, премія L’Oréal-UNESCO для жінок у науці, премія доктора Г.П. Гайнекена по біохімії та біофізиці від Нідерландської королевської академії мистецтв та наук, Міжнародна премія канадського фонду Gairdner, премія Тана Тайванської академії Sinica (2016), премія Японії, медаль Ф. Альберта Коттена за видатні хімічні дослідження (2017), премія Кавлі по нанонауці, Крунійська медаль та лекція від Лондонського королевського товариства, премія Перла Мастера Грінгарда від Університету Рокфеллера, медаль честі від Американського товариства раку (2018), премія Ніренберга (2019), премія Вольфа по медицині (2020) та ін. Дженніфер Дудна та Еммануель Шарпентьє у 2015 р. увійшли до списку ста найбільш впливових людей світу за версією журналу Time, у 2016 р. Дж. Дудна стала іноземним членом престижного Лондонського королевського товариства, а у 2018 р. увійшла в американський список 50 найбільш видатних жінок у галузі техніки за версією журналу Forbes [37].
Редагування геному людини та етичні проблеми
Перші експерименти на ссавцях
У січні 2014 року китайські учені за допомогою системи CRISPR/Cas9 модифікували геном макак. Від однієї матері народилися два мавпи, яким успішно вводили мутації в ген Ppar-γ, що відповідає за відкладення жиру, а також у ген Rag1, пов’язаний з роботою імунної системи, а саме з процесом формування різноманітності антитіл. Після успіху з мавпами все зрозуміли, що настала черга людини.
Перспективи та проблеми
І справді, дуже спокусливо, виправивши всього одну нуклеотидну основу у ДНК, звільнити людину від тяжкого спадкового захворювання, наприклад, гемофілії. Послідовність ДНК довжиною в 20 нуклеотидів, яка є «мішенню» для системи CRISPR/Cas9, як правило, не повторюється у геномі людини. Після розрізання білком Cas9 ДНК відновлюється завдяки репарації за зразком правильної копії гена з парної хромосоми. Якщо парної хромосоми немає (наприклад, при гемофілії помилка знаходиться на Х-хромосомі, яка у чоловіків тільки одна), то можна ввести в клітину разом з crРНК та білком Cas9 фрагмент з правильною послідовністю ДНК потрібного гена. Крім того, за допомогою CRISPR/Cas9 можна вимикати окремі гени та ідентифікувати ті, які відповідають за виживання клітин при раку. І виявивши в окремих клітинах організму мутації, які призводять до розвитку раку, можна за допомогою CRISPR/Cas9 усувати їх. Для швидкого редагування будь-яких генів людини створена величезна бібліотека, що містить 73 000 різних sgRNA та охоплює 80-90% всіх послідовностей геному [41].
Етичні дилеми
Однак невідомо, як організм людини відреагує на втручання в геном. Тому можливість редагування генів людини одразу ж поставила ряд етичних проблем, особливо гострих у разі редагування генів ембріона людини. Мають ли право учені або батьки майбутньої дитини приймати рішення про зміну генів ембріона, про зміну сутності людини, яка повинна народитися? Адже ніхто не запитує згоди цієї людини, тому що вона ще не існує. Де гарантія, що рішення, прийняті зараз, виявляться такими розумними, якими вони здаються зараз? Готові ли учені взяти на себе відповідальність за непередбачені наслідки таких експериментів? І що взагалі станеться з людиною, якщо вона почне змінювати свої гени на свій розсуд? Ці запитання зараз є предметом запеклих дискусій, і поки що вони не мають однозначних відповідей.
Міжнародні рекомендації щодо редагування генів людини
Однак через надзвичайно спокусливі перспективи редагування геному людини за допомогою CRISPR/Cas9 виникли побоювання, що його можуть розпочати раніше, ніж переконатися в безпеці. Тому 24 січня 2015 року Дженніфер Дудна зібрала в долині Напа в Каліфорнії 18 авторитетних генетиків для обговорення цієї проблеми. У конференції взяв участь нобелівський лауреат Пол Берг (Paul N. Berg), який організував у 1975 році на території Державного парку-пляжу Асіломар в місті Пасіфік Гроув в Каліфорнії знамениту Асіломарську конференцію, на якій було прийнято правила роботи в генній інженерії та введено мораторій на використання технологій рекомбінантної ДНК. Після цього мораторії вводили ще двічі:
У 1997 році були проведені роботи з репродуктивного клонування людини, а у 2012 році були проведені роботи з мутагенезу вірусу птичого грипу. Результати конференції в Долині Напа були опубліковані 3 квітня 2015 року у журналі Science у вигляді меморандуму, який закликає запретити клінічні експерименти з генетичної модифікації людини, поки не будуть зрозумілі наслідки та введені правила [42]. Коментуючи висновки конференції в Напі, Джордж Черч точно зауважив, що на ній скромно замовчали головне питання: «Який же сценарій нас насправді хвилює: що методи редагування геному працюватимуть недостатньо добре або що вони, навпаки, працюватимуть дуже добре?» [43].
Евгеніка та генні мутанти
Редагування геному людини відкрило теоретичну можливість не тільки лікувати генетичні захворювання, але й створювати людей з бажаними рисами. Одразу відродилися забуті після Другої світової війни ідеї створення «надлюдини» та ідеї евгеніки — вчення про поліпшення людської породи, яке було запропоновано Френсісом Гальтоном (Francis Galton), двоюрідним братом Чарльза Дарвіна. І справді, чому б не створити досконаліших людей: розумніших, красивіших, сильніших? Хоча при цьому неминуче виникають ідеї створення людей з певним призначенням, наприклад, ідеальних солдатів: сильних, витривалих, без почуття болю та страху. Страшно подумати, які можуть бути наслідки таких фантастичних можливостей науки. Тому, звичайно, люди ставляться до успіхів генетики з обережністю.
На жаль, а може й на щастя, ми не маємо достатніх знань щодо генетики складних людських ознак. Можливо, генетика взагалі не грає вирішальну роль у формуванні таких ознак. Однак багато хто бачить у технології CRISPR/Cas9 перший крок до створення нової раси людей — генних мутантів, передбачають появу зомбі, змішання видів та всесвітній генний апокаліпсис.
Безпека та потенціальні ризики
Бiологічна зброя та національна безпека
У доповіді з оцінки світових загроз для США від 9 лютого 2016 року директор національної розвідки Джеймс Р. Клаппер (James R. Clapper) назвав редагування геному потенціальною зброєю масового знищення, оскільки біологічні агенти або продукти, створені в країнах з нормативними або етичними стандартами, «відмінними від західних», можуть виявитися шкідливими. У документі стверджується, що розповсюдження, низька вартість та прискорені темпи розвитку технології CRISPR/Cas9, навмисне або ненавмисне зловживання нею можуть призвести до далекосяжних наслідків для економічної та національної безпеки США [44].
Біохакінг та домашні експерименти
Одночасно простота технології CRISPR/Cas9 та доступність у США реактивів для її реалізації дали можливість біохакерам втручатися в геном живих організмів у домашніх умовах, не маючи особливих навичок та обладнання. Так, колишній учений NASA Джозая Зайнер (Josiah Zayner), який має кандидатський ступень із біохімії Чиказького університету, заявив, що він є першою людиною, яка спробувала модифікувати свій власний геном за допомогою інноваційної технології редагування генів CRISPR. За словами Зайнера, він почав експериментувати з CRISPR у своєму гаражі влітку 2016 року, вводячи собі ген GFP, який викликає світіння медуз. Сам він не почав світитися, але біопсія показала, що новий ген присутній в його клітинах. Компанія Зайнера The Odin займається продажем комплектів «Змініть свої гени самі» вартістю $20, однак FDA заборонила розповсюдження цього продукту, назвавши його шахрайством. Тепер біохакер пропонує клієнтам купувати інструменти для генного редагування рослин та тварин. Таких випадків досить багато: підлітки, студенти, генетики-любителі намагалися застосовувати технологію CRISPR для того, щоб збільшити власні м’язи, позбутися герпесу або вилікувати СНІД, однак ці спроби були безуспішними. Неконтрольоване розповсюдження технології CRISPR являє значну небезпеку через можливість створення біологічної зброї та загрозу біотероризму.
Дослідники з Університету Альберти в Едмонтоні (Канада) відтворили з нуля вимерлий вірус чуми. Вони придбали у комерційної компанії фрагменти ДНК, що перекриваються, з’єднали їх разом та ввели в клітини, інфіковані іншим типом поксвірусу (оспи).
Робота тривала півроку та коштувала приблизно $100 тис. Цей експеримент поставив край десятирічним дебатам про те, чи потрібно знищити два зразки чуми у світі: в Центрі контролю та профілактики захворювань в Атланті (США) та у Державному науковому центрі вірусології та біотехнології «Вектор» в Кольцові під Новосибірськом (РФ), так як він довів, що учені, які хочуть експериментувати з вірусом чуми, можуть створити його самостійно. Подібні роботи повинні проводитися під суворим контролем з боку держави, але це важко зробити, так як значна частина наукових досліджень здійснюється за рахунок грошей комерційних організацій, а фінансування легко можна залучити через веб-сайти краудфондингу. Клоновані фрагменти ДНК можна замовити через спеціальні інтернет-сайти (наприклад, Science Exchange), однак невдовзі в цьому не буде необхідності, так як на столі в кожного біохакера буде стояти принтер для синтезу ДНК BioXp 3200, який продається приблизно за $65 тис. та нагадує струменевий принтер, в якому замість палітри кольорів CMYK використовуються букви генетичного коду — AGTC. А свої домашні експерименти біохакери можуть розпочати з ДНК-платформи від Amino Labs, генетичної печі Easy Bake, яка коштує менше, ніж iPad, або з набору для редагування генів CRISPR від The Odin за $159 [45].
Список использованной литературы [REFERENCES]
1. Meselson M., Yuan R. DNA restriction enzyme from E. coli. Nature. 1968. 217(5134): 1110–1114. DOI: https://doi. org/10.1038/2171110a0
2. Weiss B., Richardson C.C. Enzymatic breakage and joining of deoxyribonucleic acid, I. Repair of single-strand breaks in DNA by an enzyme system from Escherichia coli infected with T4 bacteriophage. Proc. Natl. Acad. Sci. USA. 1967. 57(4): 1021–1028. DOI: https://doi.org/10.1073/pnas.57.4.1021
3. Deltcheva E., Chylinski K., Sharma C.M., Gonzales K., Chao Y., Pirzada Z.A., Eckert M.R., Vogel J., Charpentier E. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III. Nature. 2011. 471(7340): 602– 607. DOI: https://doi.org/10.1038/nature09886
4. Westra E.R., Semenova E., Datsenko K.A., Jackson R.N., Wiedenheft B., Severinov K., Brouns S.J. Type I-E CRISPR-cas systems discriminate target from non-target DNA through base pairing-independent PAM recognition. PLoS Genet. 2013. 9(9): e1003742. DOI: https://doi.org/10.1371/journal.pgen.1003742
5. Ishino Y., Shinagawa H., Makino K., Amemura M., Nakata A. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J. Bacteriol. 1987. 169(12): 5429–5433. DOI: https://doi.org/10.1128/JB.169.12.5429-5433.1987
6. Nakata A., Amemura M., Makino K. Unusual nucleotide arrangement with repeated sequences in the Escherichia coli K-12 chromosome. J. Bacteriol. 1989. 171(6): 3553–3556. DOI: https://doi.org/10.1128/JB.171.6.3553-3556.1989
7. Groenen P.M., Bunschoten A.E., van Soolingen D., van Embden J.D. Nature of DNA polymorphism in the direct repeat cluster of Mycobacterium tuberculosis; application for strain differentiation by a novel typing method. Mol. Microbiol. 1993. 10(5): 1057–1065. DOI: https://doi.org/10.1111/j.1365-2958.1993.tb00976.x
8. Mojica F.J., Díez-Villaseñor C., Soria E., Juez G. Biological significance of a family of regularly spaced repeats in the genomes of archaea, bacteria and mitochondria. Mol. Microbiol. 2000. 36(1): 244–246. DOI: https://doi.org/10.1046/ j.1365-2958.2000.01838.x
9. Jansen R., Embden J.D., Gaastra W., Schouls L.M. Identification of genes that are associated with DNA repeats in prokaryotes. Mol. Microbiol. 2002. 43(6): 1565–1575. DOI: https://doi.org/10.1046/j.1365-2958.2002.02839.x
10. Mojica F.J., Díez-Villaseñor C., García-Martínez J., Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. Journal of Molecular Evolution. 2005. 60(2): 174–182. DOI: https://doi. org/10.1007/s00239-004-0046-3
11. Pourcel C., Salvignol G., Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies. Microbiology. 2005. 151(3): 653–663. DOI: https://doi.org/10.1099/mic.0.27437-0
12. Bolotin A., Quinquis B., Sorokin A., Ehrlich S.D. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin. Microbiology. 2005. 151(8): 2551–2561. DOI: https://doi. org/10.1099/mic.0.28048-0
13. Barrangou R., Fremaux C., Deveau H., Richards M., Boyaval P., Moineau S., Romero D.A., Horvath P. CRISPR provides acquired resistance against viruses in prokaryotes. Science. 2007. 315(5819): 1709–1712. DOI: https://doi. org/10.1126/science.1138140
14. Brouns S.J., Jore M.M., Lundgren M., Westra E.R., Slijkhuis R.J., Snijders A.P., Dickman M.J., Makarova K.S., Koonin E.V., van der Oost J. Small CRISPR RNAs guide antiviral defense in prokaryotes. Science. 2008. 321(5891): 960–964. DOI: https://doi.org/10.1126/science.1159689
15. Marraffini L.A., Sontheimer E.J. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA. Science. 2008. 322(5909): 1843–1845. DOI: https://doi.org/10.1126/science.1165771
16. Sontheimer E., Marraffini L. Target DNA interference with crRNA. U.S. Provisional Patent Application 61/009, 317, filed September 23, 2008; later published as US2010/0076057 (abandoned).
17. Jinek M., Chylinski K., Fonfara I., Hauer M., Doudna J.A, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 2012. 337(6096): 816–821. DOI: https://doi.org/10.1126/ science.1225829
18. Gasiunas G., Barrangou R., Horvath P., Siksnys V. Cas9-crRNA ribonucleoprotein complex mediates specific DNA cleavage for adaptive immunity in bacteria. Proc. Natl. Acad. Sci. USA. 2012. 109: E2579–E2586. DOI: https://doi. org/10.1073/pnas.1208507109
19. Mali P., Yang L., Esvelt K.M., Aach J., Guell M., DiCarlo J.E., Norville J.E., Church G.M. RNA-guided human genome engineering via Cas9. Science. 2013. 339(6121): 823–826. DOI: https://doi.org/10.1126/science.1232033
20. Cong L., Ran F.A., Cox D., Lin S., Barretto R., Habib N., Hsu P.D., Wu X., Jiang W., Marraffini L.A., Zhang F. Multiplex genome engineering using CRISPR/Cas systems. Science. 2013. 339(6121): 819–823. DOI: https://doi. org/10.1126/science.1231143
21. Cho S.W., Kim S., Kim J.M., Kim J.S. Targeted genome engineering in human cells with the Cas9 RNA-guided endonuclease. Nat. Biotechnol. 2013. 31(3): 230–232. DOI: https://doi.org/10.1038/nbt.2507
22. Meganuclease. Wikipedia. https://en.wikipedia.org/wiki/Meganuclease
23. O’Connell M.R., Oakes B.L., Sternberg S.H., East-Seletsky A., Kaplan M., Doudna J.A. Programmable RNA recognition and cleavage by CRISPR/Cas9. Nature. 2014. 516(7530): 263–266. DOI: https://doi.org/10.1038/nature13769
24. Nelles D.A., Fang M.Y., O’Connell M.R., Xu J.L., Markmiller S.J., Doudna J.A., Yeo G.W. Programmable RNA tracking in live cells with CRISPR/Cas9. Cell. 2016. 165(2): 488–496. DOI: https://doi.org/10.1016/j.cell.2016.02.054
25. Vandenberghe L.H. Addgene: molecular therapy interview with Melina Fan and Karen Guerin. https://www. cell.com/molecular-therapy-family/molecular-therapy/fulltext/S1525-0016(18)30582-3 DOI: https://doi. org/10.1016/j.ymthe.2018.12.001
26. Brown K.V. Why CRISPR-edited food may be in supermarkets sooner than you think. https://gizmodo.com/whycrispr-edited-food-may-be-in-supermarkets-sooner-th-1822025033
27. Lee J., Wang F. Gene-edited baby by Chinese scientist: the opener of the pandora’s box. Science Insights. 2018. 2018:e000178. DOI: https://doi.org/10.15354/si.18.co015
28. Reardon S. CRISPR gene-editing creates wave of exotic model organisms. Nature. 2019. 568(7753): 441–442. DOI: https://doi.org/10.1038/d41586-019-01300-9
29. Wade N. Genes color a butterfly’s wings. Now scientists want to do it themselves. https://www.nytimes. com/2017/09/18/science/butterfly-wing-color-patterns-gene-editing.html
30. Qi L.S., Larson M.H., Gilbert L.A., Doudna J.A., Weissman J.S., Arkin A.P., Lim W.A. Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression. Cell. 2013. 152(5): 1173–1183. DOI: https:// doi.org/10.1016/j.cell.2013.02.022
31. Kungulovski G., Jeltsch A. Epigenome editing: state of the art, concepts, and perspectives. Trends Genet. 2016. 32(2): 101–113. DOI: https://doi.org/10.1016/j.tig.2015.12.001
32. Pefanis E., Wang J.G., Rothschild G., Lim J., Kazadi D., Sun J.B., Federation A., Chao J., Elliott O., Liu Z.P., Economides A.N., Bradner J.E., Rabadan R., Basu U. RNA exosome-regulated long non-coding RNA transcription controls super-enhancer activity. Cell. 2015. 161(4): 774–789. DOI: https://doi.org/10.1016/j.cell.2015.04.034
33. Elling R., Chan J., Fitzgerald K.A. Emerging role of long noncoding RNAs as regulators of innate immune cell development and inflammatory gene expression. Eur. J. Immunol. 2016. 46(3): 504–512. DOI: https://doi.org/10.1002/ eji.201444558
34. Chen B., Gilbert L.A., Cimini B.A., Schnitzbauer J., Zhang W., Li G.W., Park J., Blackburn E.H., Weissman J.S., Qi L.S., Huang B. Dynamic imaging of genomic loci in living human cells by an optimized CRISPR/Cas system. Cell. 2013. 155(7): 1479–1491. DOI: https://doi.org/10.1016/j.cell.2013.12.001
35. Hajian R., Balderston S., Tran T., deBoer T., Etienne J., Sandhu M., Wauford N.A., Chung J.Y., Nokes J., Athaiya M., Paredes J., Peytavi R., Goldsmith B., Murthy N., Conboy I.M., Aran K. Detection of unamplified target genes via CRISPR-Cas9 immobilized on a graphene field-effect transistor. Nat. Biomed. Eng. 2019. 3(6): 427–437. DOI: https://doi.org/10.1038/s41551-019-0371-x
36. CRISPR’s future for point-of-care diagnostics. https://www.diagnosticsworldnews.com/news/2020/02/18/ crispr%27s-future-for-point-of-care-diagnostics
37. List of awards and honors received by Jennifer Doudna. Wikipedia. https://en.wikipedia.org/wiki/List_of_awards_ and_honors_received_by_Jennifer_Doudna
38. Niu Y., Shen B., Cui Y., Chen Y., Wang J., Wang L., Kang Y., Zhao X., Si W., Li W., Xiang A.P., Zhou J., Guo X., Bi Y., Si C., Hu B., Dong G., Wang H., Zhou Z., Li T., Tan T., Pu X., Wang F., Ji S., Zhou Q., Huang X., Ji W., Sha J. Generation of gene-modified cynomolgus monkey via Cas9/RNA-mediated gene targeting in one-cell embryos. Cell. 2014. 156(4): 836–843. DOI: https://doi.org/10.1016/j.cell.2014.01.027
39. Shalem O., Sanjana N.E., Hartenian E., Shi X., Scott D.A., Mikkelsen T.S., Heckl D., Ebert B.L., Root D.E., Doench J.G., Zhang F. Genome-scale CRISPR/Cas9 knockout screening in human cells. Science. 2014. 343(6166): 84–87. DOI: https://doi.org/10.1126/science.1247005
40. Raphael B.J., Dobson J.R., Oesper L., Vandin F. Identifying driver mutations in sequenced cancer genomes: computational approaches to enable precision medicine. Genome Med. 2014. 6(1): 5. DOI: https://doi.org/10.1186/gm524
41. Wang T., Wei J.J., Sabatini D.M., Lander E.S. Genetic screens in human cells using the CRISPR/Cas9 system. Science. 2014. 343(6166): 80–84. DOI: https://doi.org/10.1126/science.1246981
42. Baltimore D., Berg P., Botchan M., Carroll D., Charo R.A., Church G., Corn J.E., Daley G.Q., Doudna J.A., Fenner M., Greely H.T., Jinek M., Martin G.S., Penhoet E., Puck J., Sternberg S.H., Weissman J.S., Yamamoto K.R. Biotechnology. A prudent path forward for genomic engineering and germline gene modification. Science. 2015. 348(6230): 36–38. DOI: https://doi.org/10.1126/science.aab1028
43. Vogel G. Bioethics. Embryo engineering alarm. Science. 2015. 347(6228): 1301. DOI: https://doi.org/10.1126/science.347.6228.1301
44. Clapper J.R. Worldwide threat assessment of the US intelligence community. https://www.dni.gov/files/documents/SASC_Unclassified_2016_ATA_SFR_FINAL.pdf
45. Baumgaertner E. As D.I.Y. gene editing gains popularity, ‘Someone is going to get hurt’. https://www.nytimes. com/2018/05/14/science/biohackers-gene-editing-virus.html
46. Liang P., Xu Y., Zhang X., Ding C., Huang R., Zhang Z., Lv J., Xie X., Chen Y., Li Y., Sun Y., Bai Y., Songyang Z., Ma W., Zhou C., Huang J. CRISPR/Cas9-mediated gene editing in human tripronuclear zygotes. Protein Cell. 2015. 6(5): 363–372. DOI: https://doi.org/10.1007/s13238-015-0153-5
47. Ran F.A., Hsu P.D., Lin C.Y., Gootenberg J.S., Konermann S., Trevino A.E., Scott D.A., Inoue A., Matoba S., Zhang Y., Zhang F. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell. 2013. 154(6): 1380–1389. DOI: https://doi.org/10.1016/j.cell.2013.08.021
48. Fu Y., Sander J.D., Reyon D., Cascio V.M., Joung J.K. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. Nat. Biotechnol. 2014. 32(3): 279–284. DOI: https://doi.org/10.1038/nbt.2808
49. Kleinstiver B.P., Prew M.S., Tsai S.Q., Topkar V.V., Nguyen N.T., Zheng Z., Gonzales A.P., Li Z., Peterson R.T., Yeh J.R., Aryee M.J., Joung J.K. Engineered CRISPR/Cas9 nucleases with altered PAM specificities. Nature. 2015. 523(7561): 481–485. DOI: https://doi.org/10.1038/nature14592
50. Guilinger J.P., Thompson D.B., Liu D.R. Fusion of catalytically inactive Cas9 to FokI nuclease improves the specificity of genome modification. Nat. Biotechnol. 2014. 32(6): 577–582. DOI: https://doi.org/10.1038/nbt.2909
51. Slaymaker I.M., Gao L., Zetsche B., Scott D.A., Yan W.X., Zhang F. Rationally engineered Cas9 nucleases with improved specificity. Science. 2016. 351(6268): 84–88. DOI: https://doi.org/10.1126/science.aad5227
52. Kleinstiver B.P., Pattanayak V., Prew M.S., Tsai S.Q., Nguyen N.T., Zheng Z., Joung J.K. High-fidelity CRISPR/Cas9 nucleases with no detectable genome-wide off-target effects. Nature. 2016. 529(7587): 490–495. DOI: https://doi. org/10.1038/nature16526
53. Zhou W., Deiters A. Conditional Control of CRISPR/Cas9 Function. Angew. Chem. Int. Ed. Engl. 2016. 55(18): 5394–5399. DOI: https://doi.org/10.1002/anie.201511441
54. Byrne J.A., Pedersen D.A., Clepper L.L., Nelson M., Sanger W.G., Gokhale S., Wolf D.P., Mitalipov S.M. Producing primate embryonic stem cells by somatic cell nuclear transfer. Nature. 2007. 450(7169): 497–502. DOI: https://doi. org/10.1038/nature06357
55. Tachibana M., Sparman M., Sritanaudomchai H., Ma H., Clepper L., Woodward J., Li Y., Ramsey C., Kolotushkina O., Mitalipov S. Mitochondrial gene replacement in primate offspring and embryonic stem cells. Nature. 2009. 461(7262): 367–372. DOI: https://doi.org/10.1038/nature08368
56. Tachibana M., Amato P., Sparman M., Gutierrez N.M., Tippner-Hedges R., Ma H., Kang E., Fulati A., Lee H.S., Sritanaudomchai H., Masterson K., Larson J., Eaton D., Sadler-Fredd K., Battaglia D., Lee D., Wu D., Jensen J., Patton P., Gokhale S., Stouffer R.L., Wolf D., Mitalipov S. Human embryonic stem cells derived by somatic cell nuclear transfer. Cell. 2013. 153(6): 1228–1238. DOI: https://doi.org/10.1016/j.cell.2013.06.042
57. Kang E., Wu J., Gutierrez N.M., Koski A., Tippner-Hedges R., Agaronyan K., Platero-Luengo A., Martinez-Redondo P., Ma H., Lee Y., Hayama T., Van Dyken C., Wang X., Luo S., Ahmed R., Li Y., Ji D., Kayali R., Cinnioglu C., Olson S., Jensen J., Battaglia D., Lee D., Wu D., Huang T., Wolf D.P., Temiakov D., Belmonte J.C., Amato P., Mitalipov S. Mitochondrial replacement in human oocytes carrying pathogenic mitochondrial DNA mutations. Nature. 2016. 540(7632): 270–275. DOI: https://doi.org/10.1038/nature20592
58. Ma H., Marti-Gutierrez N., Park S.W., Wu J., Lee Y., Suzuki K., Koski A., Ji D., Hayama T., Ahmed R., Darby H., Van Dyken C., Li Y., Kang E., Park A.R., Kim D., Kim S.T., Gong J., Gu Y., Xu X., Battaglia D., Krieg S.A., Lee D.M., Wu D.H., Wolf D.P., Heitner S.B., Belmonte J.C.I., Amato P., Kim J.S., Kaul S., Mitalipov S. Correction of a pathogenic gene mutation in human embryos. Nature. 2017. 548(7668): 413–419. DOI: https://doi.org/10.1038/nature23305
59. Second woman carrying gene-edited baby, Chinese authorities confirm. https://www.theguardian.com/science/2019/jan/22/second-woman-carrying-gene-edited-baby-chinese-authorities-confirm
60. CRISPR scientist gets three years of jail time for creating gene-edited babies. https://gizmodo.com/crispr-scientistgets-three-years-of-jail-time-for-crea-1840724277
61. Act now on CRISPR babies. Nature. 2019. 570(137). DOI: https://doi.org/10.1038/d41586-019-01786-3
62. Collins F.S. NIH Director on Human Gene Editing: ‘We Must Never Allow our Technology to Eclipse our Humanity’. https://www.discovermagazine.com/health/nih-director-on-human-gene-editing-we-must-never-allow-ourtechnology-to
63. Gene mutation meant to protect from HIV ‘raises risk of early death’. https://www.theguardian.com/science/2019/ jun/03/gene-mutation-protect-hiv-raises-risk-early-death
64. Andorno R., Yamin A.E. The right to design babies? Human rights and bioethics. https://www.openglobalrights.org/ the-right-to-design-babies-human-rights-and-bioethics/
65. Citorik R.J., Mimee M., Lu T.K. Sequence-specific antimicrobials using efficiently delivered RNA-guided nucleases. Nat. Biotechnol. 2014. 32(11): 1141–1145. DOI: https://doi.org/10.1038/nbt.3011
66. Bikard D., Euler C.W., Jiang W., Nussenzweig P.M., Goldberg G.W., Duportet X., Fischetti V.A., Marraffini L.A. Exploiting CRISPR-Cas nucleases to produce sequence-specific antimicrobials. Nat. Biotechnol. 2014. 32(11): 1146– 1150. DOI: https://doi.org/10.1038/nbt.3043
67. Yosef I., Manor M., Kiro R., Qimron U. Temperate and lytic bacteriophages programmed to sensitize and kill antibiotic-resistant bacteria. Proc. Natl. Acad. Sci. USA. 2015. 112(23): 7267–7272. DOI: https://doi.org/10.1073/ pnas.1500107112
68. Gantz V.M., Jasinskiene N., Tatarenkova O., Fazekas A., Macias V.M., Bier E., James A.A. Highly efficient Cas9-mediated gene drive for population modification of the malaria vector mosquito Anopheles stephensi. Proc. Natl. Acad. Sci. USA. 2015. 112(49): E6736–E6743. DOI: https://doi.org/10.1073/pnas.1521077112
69. Gantz V.M., Bier E. The mutagenic chain reaction: a method for converting heterozygous to homozygous mutations. Science. 2015. 348(6233): 442–444. DOI: https://doi.org/10.1126/science.aaa5945
70. Stokstad E. Genetically engineered moths can knock down crop pests, but will they take off? https://www.sciencemag.org/news/2020/01/genetically-engineered-moths-can-knock-down-crop-pests-will-they-take DOI: https://doi.org/10.1126/science.abb1078
71. Yang L., Güell M., Niu D., George H., Lesha E., Grishin D., Aach J., Shrock E., Xu W., Poci J., Cortazio R., Wilkinson R.A., Fishman J.A., Church G. Genome-wide inactivation of porcine endogenous retroviruses (PERVs). Science. 2015. 350(6264): 1101–1104. DOI: https://doi.org/10.1126/science.aad1191
72. Niu D., Wei H.J., Lin L., George H., Wang T., Lee I.H., Zhao H.Y., Wang Y., Kan Y., Shrock E., Lesha E., Wang G., Luo Y., Qing Y., Jiao D., Zhao H., Zhou X., Wang S., Wei H., G ell M., Church G.M., Yang L. Inactivation of porcine endogenous retrovirus in pigs using CRISPR-Cas9. Science. 2017. 357(6357): 1303–1307. DOI: https://doi. org/10.1126/science.aan4187
73. Gene editing spurs hope for transplanting pig organs into humans. https://www.nytimes.com/2017/08/10/health/ gene-editing-pigs-organ-transplants.html
74. Nunes Dos Santos R.M., Carneiro D’Albuquerque L.A., Reyes L.M., Estrada J.L., Wang Z.Y., Tector M., Tector A.J. CRISPR/Cas and recombinase-based human-to-pig orthotopic gene exchange for xenotransplantation. J. Surg. Res. 2018. 229: 28–40. DOI: https://doi.org/10.1016/j.jss.2018.03.051
75. Dong C., Qu L., Wang H., Wei L., Dong Y., Xiong S. Targeting hepatitis B virus cccDNA by CRISPR/Cas9 nuclease efficiently inhibits viral replication. Antiviral Res. 2015. 118: 110–117. DOI: https://doi.org/10.1016/j. antiviral.2015.03.015
76. Kaminski R., Chen Y., Fischer T., Tedaldi E., Napoli A., Zhang Y., Karn J., Hu W., Khalili K. Elimination of HIV-1 genomes from human T-lymphoid cells by CRISPR/Cas9 gene editing. Sci. Rep. 2016. 6: 22555. DOI: https://doi. org/10.1038/srep22555
77. Wang Z., Pan Q., Gendron P., Zhu W., Guo F., Cen S., Wainberg M.A., Liang C. CRISPR/Cas9-derived mutations both inhibit HIV-1 replication and accelerate viral escape. Cell Rep. 2016. 15(3): 481–489. DOI: https://doi. org/10.1016/j.celrep.2016.03.042
78. Kang X., He W., Huang Y., Yu Q., Chen Y., Gao X., Sun X., Fan Y. Introducing precise genetic modifications into human 3PN embryos by CRISPR/Cas-mediated genome editing. J. Assist. Reprod. Genet. 2016. 33(298): 1–8. DOI: https://doi.org/10.1007/s10815-016-0710-8
79. Xu L., Yang H., Gao Y., Chen Z., Xie L., Liu Y., Liu Y., Wang X., Li H., Lai W., He Y., Yao A., Ma L., Shao Y., Zhang B., Wang C., Chen H., Deng H. CRISPR/Cas9-Mediated CCR5 Ablation in Human Hematopoietic Stem/Progenitor Cells Confers HIV-1 Resistance In Vivo. Mol Ther. 2017. 25(8): 1782–1789. DOI: https://doi.org/10.1016/j. ymthe.2017.04.027
80. Dash P.K., Kaminski R., Bella R., Su H., Mathews S., Ahooyi T.M., Chen C., Mancuso P., Sariyer R., Ferrante P., Donadoni M., Robinson J.A., Sillman B., Lin Z., Hilaire J.R., Banoub M., Elango M., Gautam N., Mosley R.L., Poluektova L.Y., McMillan J., Bade A.N., Gorantla S., Sariyer I.K., Burdo T.H., Young W.B., Amini S., Gordon J., Jacobson J.M., Edagwa B., Khalili K., Gendelman H.E. Sequential LASER ART and CRISPR treatments eliminate HIV-1 in a subset of infected humanized mice. Nat. Commun. 2019. 10(1): 2753. DOI: https://doi.org/10.1038/s41467-019- 10366-y
81. Yuan M., Webb E., Lemoine N.R., Wang Y. CRISPR-Cas9 as a powerful tool for efficient creation of oncolytic viruses. Viruses. 2016. 8(3): E72. DOI: https://doi.org/10.3390/v8030072
82. Kennedy E.M., Kornepati A.V., Goldstein M., Bogerd H.P., Poling B.C., Whisnant A.W., Kastan M.B., Cullen B.R. Inactivation of the human papillomavirus E6 or E7 gene in cervical carcinoma cells by using a bacterial CRISPR/Cas RNA-guided endonuclease. J. Virol. 2014. 88(20): 11965–11972. DOI: https://doi.org/10.1128/JVI.01879-14
83. Miller J.F., Sadelain M. The journey from discoveries in fundamental immunology to cancer immunotherapy. Cancer Cell. 2015. 27(4): 439–449. DOI: https://doi.org/10.1016/j.ccell.2015.03.007
84. Roth T.L., Puig-Saus C., Yu R., Shifrut E., Carnevale J., Li P.J., Hiatt J., Saco J., Krystofinski P., Li H., Tobin V., Nguyen D.N., Lee M.R., Putnam A.L., Ferris A.L., Chen J.W., Schickel J.N., Pellerin L., Carmody D., Alkorta-Aranburu G., Del Gaudio D., Matsumoto H., Morell M., Mao Y., Cho M., Quadros R.M., Gurumurthy C.B., Smith B., Haugwitz M., Hughes S.H., Weissman J.S., Schumann K., Esensten J.H., May A.P., Ashworth A., Kupfer G.M., Greeley S.A.W., Bacchetta R., Meffre E., Roncarolo M.G., Romberg N., Herold K.C., Ribas A., Leonetti M.D., Marson A. Reprogramming human T cell function and specificity with non-viral genome targeting. Nature. 2018. 559(7714): 405–409. DOI: https://doi.org/10.1038/s41586-018-0326-5
85. Zaroff S. CAR T-Cell therapies with a bispecific twist. Genet. Eng. Biotech. N. 2018. 38(13). https://www.genengnews.com/magazine/car-t-cell-therapies-with-a-bispecific-twist/ DOI: https://doi.org/10.1089/gen.38.13.09
86. Kojima R., Scheller L., Fussenegger M. Nonimmune cells equipped with T-cell-receptor-like signaling for cancer cell ablation. Nature Chemical Biology. 2018. 14: 42–49. DOI: https://doi.org/10.1038/nchembio.2498
87. Montel-Hagen A., Seet C.S., Li S., Chick B., Zhu Y., Chang P., Tsai S., Sun V., Lopez S., Chen H.C., He C., Chin C.J., Casero D., Crooks G.M. Organoid-induced differentiation of conventional T cells from human pluripotent stem cells. Cell Stem Cell. 2019. 24(3): 376–389.e8. DOI: https://doi.org/10.1016/j.stem.2018.12.011
88. White M.K., Khalili K. CRISPR/Cas9 and cancer targets: future possibilities and present challenges. Oncotarget. 2016. 7(11): 12305–12317. DOI: https://doi.org/10.18632/oncotarget.7104
89. Cyranoski D. CRISPR gene-editing tested in a person for the first time. Nature News. 2016. 539(7630): 479. DOI: https://doi.org/10.1038/nature.2016.20988
90. New cancer drug targets accelerate path to precision medicine. https://www.drugtargetreview.com/news/42672/ new-cancer-drug-targets-accelerate-path-to-precision-medicine/
91. Booth C., Gaspar H.B., Thrasher A.J. Treating immunodeficiency through HSC gene therapy. Trends Mol. Med. 2016. 22(4): 317–327. DOI: https://doi.org/10.1016/j.molmed.2016.02.002
92. Guan Y., Ma Y., Li Q., Sun Z., Ma L., Wu L., Wang L., Zeng L., Shao Y., Chen Y., Ma N., Lu W., Hu K., Han H., Yu Y., Huang Y., Liu M., Li D. CRISPR/Cas9-mediated somatic correction of a novel coagulator factor IX gene mutation ameliorates hemophilia in mouse. EMBO Mol. Med. 2016. 8(5): 477–488. DOI: https://doi.org/10.15252/ emmm.201506039
93. Nelson C.E., Hakim C.H., Ousterout D.G., Thakore P.I., Moreb E.A., Castellanos Rivera R.M., Madhavan S., Pan X., Ran F.A., Yan W.X., Asokan A., Zhang F., Duan D., Gersbach C.A. In vivo genome editing improves muscle function in a mouse model of Duchenne muscular dystrophy. Science. 2016. 351(6271): 403–407. DOI: https://doi.org/10.1126/ science.aad5143
94. DeWitt M.A., Magis W., Bray N.L., Wang T., Berman J.R., Urbinati F., Heo S.J., Mitros T., Mu oz D.P., Boffelli D., Kohn D.B., Walters M.C., Carroll D., Martin D.I., Corn J.E. Selection-free genome editing of the sickle mutation in human adult hematopoietic stem/progenitor cells. Sci. Transl. Med. 2016. 8(360): 360ra134. DOI: https://doi. org/10.1126/scitranslmed.aaf9336
95. CRISPR patent fight turns ugly as UC accuses Broad researchers of lying about claims. https://www.genomeweb. com/business-news/crispr-patent-fight-turns-ugly-uc-accuses-broad-researchers-lying-about-claims
96. Sanders R. UC rings out 2019 with its 20th CRISPR patent. https://news.berkeley.edu/2019/12/31/uc-rings-out2019-with-its-20th-crispr-patent/
97. Craven L., Herbert M., Murdoch A., Murphy J., Lawford Davies J., Turnbull D.M. Research into policy: a brief history of mitochondrial donation. Stem Cells. 2016. 34(2): 265–267. DOI: https://doi.org/10.1002/stem.2221
98. Callaway E. UK scientists gain license to edit genes in human embryos. Nature News. 2016. 530(7588): 18. DOI: https://doi.org/10.1038/nature.2016.19270
99. Mills P. Genome editing and human reproduction: The Nuffield Council on Bioethics’ report. https://www.bionews. org.uk/page_137343
100. Becker R. The ‘three-parent baby’ fertility doctor needs to stop marketing the procedure, FDA says. https://www. theverge.com/2017/8/5/16100680/three-parent-baby-fertility-doctor-fda-letter-violations
101. This fertility doctor is pushing the boundaries of human reproduction, with little regulation. https://www.washingtonpost.com/national/health-science/this-fertility-doctor-is-pushing-the-boundaries-of-human-reproductionwith-little-regulation/2018/05/11/ea9105dc-1831-11e8-8b08-027a6ccb38eb_story.html
102. Sangamo ZFN Technology Platform. 2018. https://www.sangamo.com/application/files/6915/3002/3307/IRTechnology_v06.12.18_1.pdf
103. Haridy R. First CRISPR therapy administered in landmark human trial. https://newatlas.com/crispr-trial-underway-vertex-gene-therapy/58643/
104. The Future of CRISPR. http://www.fwreports.com/dossier/the-future-of-crispr/#.XmgL-kFR2Uk
105. «Tegsedi»: an oligonucleotide drug against familial amyloid polyneuropathy. (in Russian). https://mosmedpreparaty.ru/news/16897 [«Тегседи»: олигонуклеотидное лекарство против семейной амилоидной полинейропатии.]
106. Stolberg S.G. The biotech death of Jesse Gelsinger. http://www.nytimes.com/1999/11/28/magazine/the-biotechdeath-of-jesse-gelsinger.html
107. Bersenev A. The history of gene therapy drugs approval on the market. http://stemcellassays.com/2011/12/history-gene-therapy-drugs-approval-market/
108. Morrison C. 1-million price tag set for Glybera gene therapy. Nature Biotechnology. 2015. 33: 217–218. DOI: https:// doi.org/10.1038/nbt0315-217
109. Kozubek J. Who will pay for CRISPR? https://www.statnews.com/2017/06/26/crispr-insurance-companies-pay/
110. Talimogene laherparepvec. Wikipedia. https://en.wikipedia.org/wiki/Talimogene_laherparepvec
111. Kegel M. Imlygic-Yervoy combo twice as effective as Yervoy in fighting melanoma, study finds. https://immunooncologynews.com/2017/10/12/melanoma-investigational-therapy-combo-imlygic-yervoy-twice-as-effective-yervoy-alone-study-finds/
112. Mullin E. A gene therapy that cures a rare genetic disease just got its first customer, a year after it was approved. http://www.businessinsider.com/gsks-strimvelis-gene-therapy-used-for-the-first-time-after-approval-2017-5
113. Al Idrus A. Orchard Therapeutics’ 2019: Pipeline progress, breaking ground on its $90M manufacturing site. https://www.fiercebiotech.com/biotech/orchard-therapeutics-2019-pipeline-progress-breaking-ground-its-90mmanufacturing-site
114. Sampson T.R., Saroj S.D., Llewellyn A.C., Tzeng Y.L., Weiss D.S. A CRISPR/Cas system mediates bacterial innate immune evasion and virulence. Nature. 2013. 497(7448): 254–257. DOI: https://doi.org/10.1038/nature12048
115. Koonin E.V., Krupovic M. Evolution of adaptive immunity from transposable elements combined with innate immune systems. Nat. Rev. Genet. 2015. 16(3): 184–192. DOI: https://doi.org/10.1038/nrg3859
116. Wright A.V., Liu J.J., Knott G.J., Doxzen K.W., Nogales E., Doudna J.A. Structures of the CRISPR genome integration complex. Science. 2017. 357(6356): 1113–1118. DOI: https://doi.org/10.1126/science.aao0679
117. Jiang F., Taylor D.W., Chen J.S., Kornfeld J.E., Zhou K., Thompson A.J., Nogales E., Doudna J.A. Structures of a CRISPR/Cas9 R-loop complex primed for DNA cleavage. Science. 2016. 351(6275): 867–871. DOI: https://doi. org/10.1126/science.aad8282
118. Zetsche B., Gootenberg J.S., Abudayyeh O.O., Slaymaker I.M., Makarova K.S., Essletzbichler P., Volz S.E., Joung J., van der Oost J., Regev A., Koonin E.V., Zhang F. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell. 2015. 163(3): 759–771. DOI: https://doi.org/10.1016/j.cell.2015.09.038
119. Gleeson A., Sawyer A. CRISPR/Cas9: the gold standard of genome editing? Biotechniques. 2018. 64(6): 239–243. DOI: https://doi.org/10.2144/btn-2018-0066
120. Sansbury B.M., Wagner A.M., Nitzan E., Gabi T., Kmeic E.B. CRISPR-directed in vitro gene editing of plasmid DNA catalyzed by Cpf1 (Cas12a) nuclease and a mammalian cell-free extract. CRISPR J. 2018. 1(2): 191–202. DOI: https://doi.org/10.1089/crispr.2018.0006
121. Burstein D., Harrington L.B., Strutt S.C., Probst A.J., Anantharaman K., Thomas B.C., Doudna J.A., Banfield J.F. New CRISPR-Cas systems from uncultivated microbes. Nature. 2017. 542(7640): 237–241. DOI: https://doi. org/10.1038/nature21059
122. Hegge J.W., Swarts D.C., van der Oost J. Prokaryotic Argonaute proteins: novel genome-editing tools? Nat. Rev. Microbiol. 2017. 16(1): 5–11. DOI: https://doi.org/10.1038/nrmicro.2017.73
123. Harrington L.B., Burstein D., Chen J.S., Paez-Espino D., Ma E., Witte I.P., Cofsky J.C., Kyrpides N.C., Banfield J.F., Doudna J.A. Programmed DNA Destruction by Miniature CRISPR-Cas14 Enzymes. Science. 2018. 362(6416): 839–842. DOI: https://doi.org/10.1126/science.aav4294
124. Abudayyeh O.O., Gootenberg J.S., Konermann S., Joung J., Slaymaker I.M., Cox D.B., Shmakov S., Makarova K.S., Semenova E., Minakhin L., Severinov K., Regev A., Lander E.S., Koonin E.V., Zhang F. C2c2 is a single-component programmable RNA-guided RNA-targeting CRISPR effector. Science. 2016. 353(6299): aaf5573. DOI: https://doi. org/10.1126/science.aaf5573
125. Smargon A.A., Cox D.B., Pyzocha N.K., Zheng K., Slaymaker I.M., Gootenberg J.S., Abudayyeh O.A., Essletzbichler P., Shmakov S., Makarova K.S., Koonin E.V., Zhang F. Cas13b is a type VI-B CRISPR-associated RNAguided RNase differentially regulated by accessory proteins Csx27 and Csx28. Mol. Cell. 2017. 65(4): 618–630.e7. DOI: https://doi.org/10.1016/j.molcel.2016.12.023
126. Yan W.X., Chong S., Zhang H., Makarova K.S., Koonin E.V., Cheng D.R., Scott D.A. Cas13d is a compact RNAtargeting type VI CRISPR effector positively modulated by a WYL-domain-containing accessory protein. Mol Cell. 2018. 70(2): 327–339. DOI: https://doi.org/10.1016 / j.molcel.2018.02.028
127. Chatterjee P., Jakimo N., Jacobson J.M. Minimal PAM specificity of a highly similar SpCas9 ortholog. Sci. Adv. 2018. 4(10): eaau0766. DOI: https://doi.org/10.1126/sciadv.aau0766
128. New DNA ‘shredder’ technique goes beyond CRISPR’s scissors. https://www.drugtargetreview.com/news/42518/ new-dna-shredder-technique-goes-beyond-crisprs-scissors/
129. Sadhu M.J., Bloom J.S., Day L., Siegel J.J., Kosuri S., Kruglyak L. Highly parallel genome variant engineering with CRISPR-Cas9. Nat. Genet. 2018. 50(4): 510–514. DOI: https://doi.org/10.1038/s41588-018-0087-y
130. Enzyme fragment complementation assay technology. https://www.discoverx.com/technologies-platforms/enzyme-fragment-complementation-technology
131. Biosensor development using CRISPR to quantify endogenous protein modulated by targeted protein degraders. http://www.healthtech.com/eurofins-biosensor-Development-using-crispr/
132. Zengerle M., Chan K.-H., Ciulli A. Selective small molecule induced degradation of the BET bromodomain protein BRD4. ACS Chem. Biol. 2015. 10(8): 1770. DOI: https://doi.org/10.1021/acschembio.5b00216
133. Single-stranded DNA synthesis service. https://www.genscript.com/new-single-stranded-dna-synthesis-service. html
134. Strecker J., Ladha A., Gardner Z., Schmid-Burgk J.L., Makarova K.S., Koonin E.V., Zhang F. RNA-guided DNA insertion with CRISPR-associated transposases. Science. 2019. 365(6448): 48–53. DOI: https://doi.org/10.1126/ science.aax9181
135. Stafforst T., Schneider M.F. An RNA-deaminase conjugate selectively repairs point mutations. Angew. Chem. Int. Ed. Engl. 2012. 51(44): 11166–11169. DOI: https://doi.org/10.1002/anie.201206489
136. Reardon S. Step aside CRISPR, RNA editing is taking off. Nature. 2020. 578(7793): 24–27. DOI: https://doi. org/10.1038/d41586-020-00272-5 137. Pennisi E. The CRISPR craze. Science. 2013. 341(6148): 833–836. DOI: https://doi.org/10.1126/science.341. 6148.833
138. Gene editing like CRISPR is too important to be left to scientists alone. https://www.theguardian.com/commentisfree/2019/oct/22/gene-editing-crispr-scientists